Практикум 8
Задание 1
Я выбрал скаффолд NW_026558161.Его длинна 29299 bp но участок с меньшей длинной, содержащий CDS мне найти не удалось.Он содержит один ген LOC129232986.Координаты CDS в этой записи 18568..18680,19239..19336 на - цепи ДНК. Получается 18681..19238 это интрон.
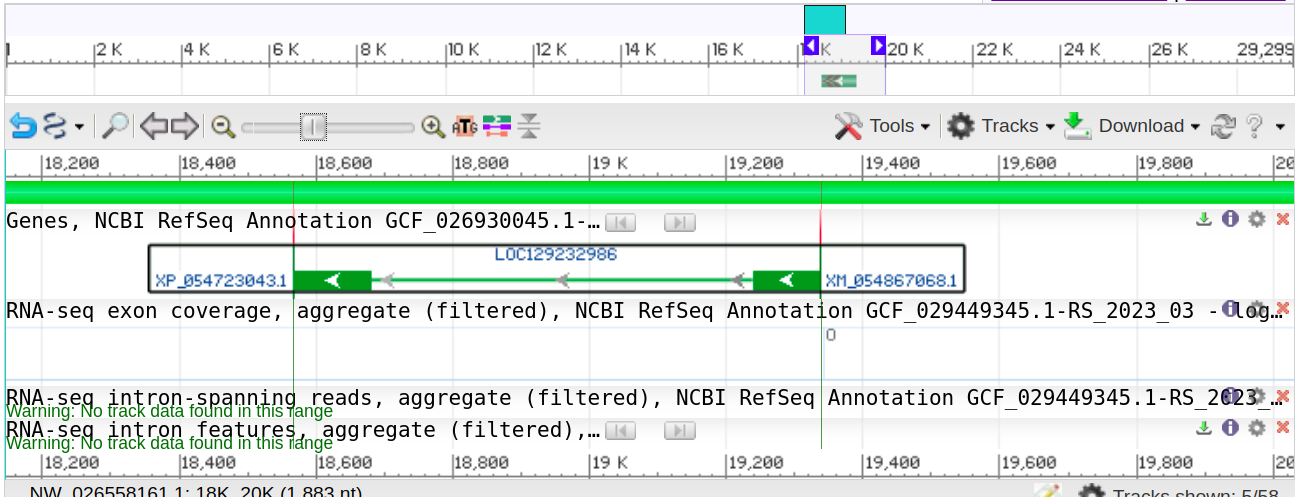 Рис 1. Схема расположения гена в скаффолде NW_026558161.В разделе Genes мы видим схему гена LOC129232986.Светло-зеленым
показан интрон, темно-зелеными прямоугольниками показаны два экзона.После сплайсинга они сошьются в одну мРНК.
Рис 1. Схема расположения гена в скаффолде NW_026558161.В разделе Genes мы видим схему гена LOC129232986.Светло-зеленым
показан интрон, темно-зелеными прямоугольниками показаны два экзона.После сплайсинга они сошьются в одну мРНК.
Далее я скачал скаффолд в fasta формате
Далее с этой последовательностью я запускал BLAST на NCBI. Я искал по классу насекомые(Insecta) с Taxonomy ID: 50557.Классы пауки(Arachnida) и насекомые(Insecta) относятся к типу членистоногие(Arthropoda).
blastn:Word size:15.E-value 0.001 . Весь день выдает ошибку,пробовал крутить параметры, не помогает
megablast:Word size:28. E-value 0.001
blastx:Word size:5.E-value 0.001 Получено не менее 100 находок.Здесь e-value у всех находок еще выше, чем у megablast.Также видно разнообразие организмов.
tblastx:Word size:3.E-value 0.001 Ошибка.Пишет что я превысил лимит использования процессора.
blastn применяется если если известна нуклеотидная последовательность(она и будет запросом) и по ней хочется найти похожие последовательности в нуклеотидных базах данных.Пример: получена неизвестная нуклеотидная последовательность и надо найти ей гомологов.
megablast подвид blastn только более быстрый и применим для последовательностей про которые заранее известно что они схожи.Пример: установление родства между людьми.
blastx принимает на вход нуклеотидную последовательность, транслирует ее в 6 рамках. И ищет по белковой базе данных.Пример: проверить является ли данная последовательность белок-кодирующей при условии что известны все белки данного организма.
tblastx принимает на вход нуклеотидную последовательность, транслирует ее в 6 рамках и ещет по базе данных полученной транслированием нуклеотидной базы данных. Пример: Известно что данная нуклеотидная последовательность белок-кодирующая, но организм из которого получен белок не известен.Поиск по blastx не дал результатов, тогда для поиска родственников можно попробовать применить tblastx.
Задание 2
Скачал BLAST себе на ноутбук.Мой файл с fasta последовательностью называется GCF_026930045.1_Udiv.v.3.1_genomic.fna
Создаю локальную базу командой:
./bin/makeblastdb -in GCA_026930045.1_Udiv.v.3.1_genomic.fna -dbtype nucl
Разделяю файл и рибосомой на 2 фаста-файла 16S.fasta и 23S.fasta.И провожу по каждой локальный поиск../bin/blastn -task blastn -query 16S.fasta -db GCA_026930045.1_Udiv.v.3.1_genomic.fna -outfmt 7
./bin/blastn -task blastn -query 23S.fasta -db GCA_026930045.1_Udiv.v.3.1_genomic.fna -outfmt 7
2. blastn т.к. работа производилась с нуклеотидной последовательностью.
3. У 16S 7 находок.Среди них гомологов не обнаружено.У 23S обнаружены гомолог: ген рРНК LOC129232985
4. Аннотация у LOC129232985 есть это 5.8S рРНК.